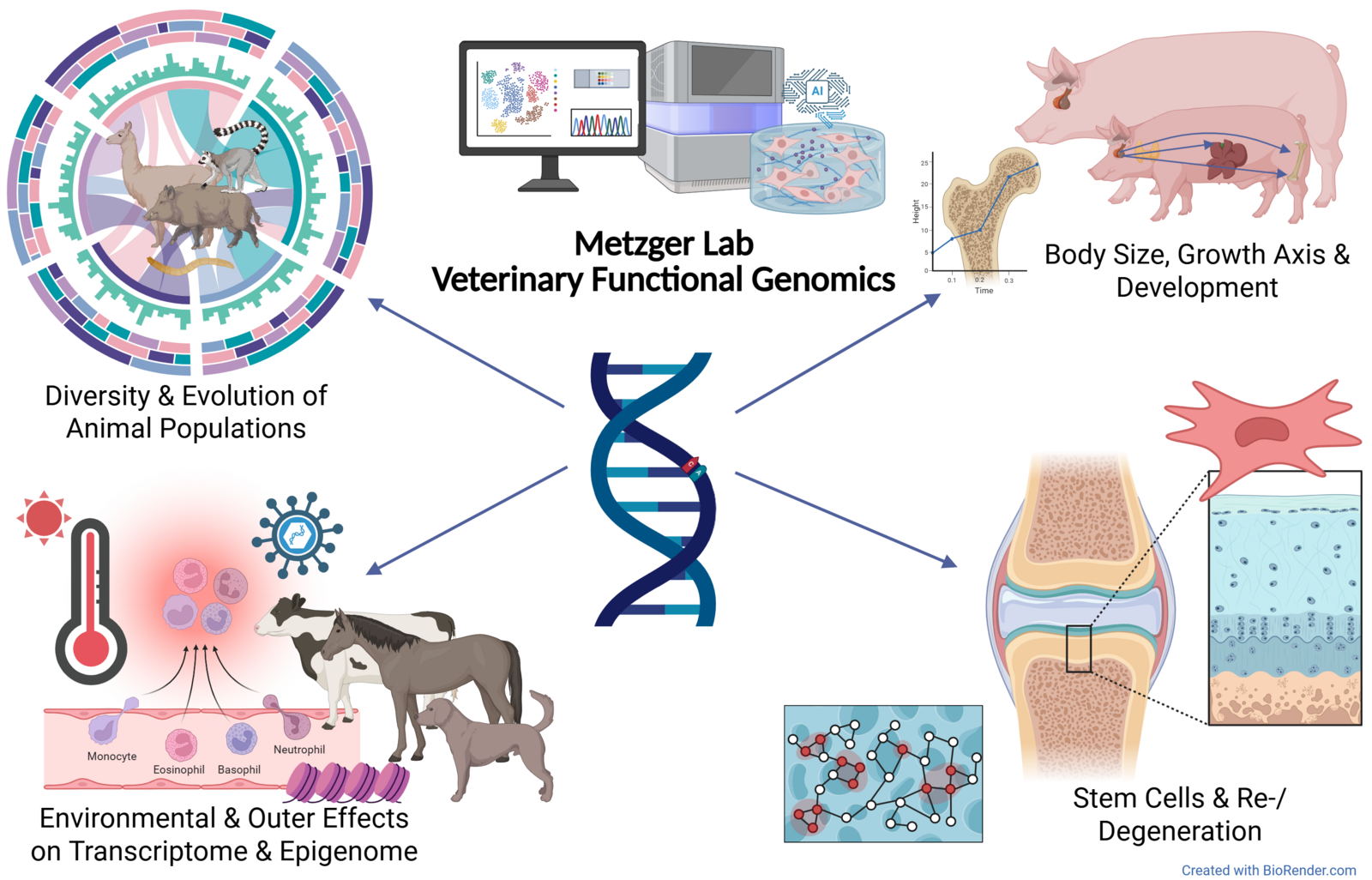

Inhalte unserer Forschung
Unsere Arbeitsgruppe beschäftigt sich mit der Variation und Architektur des Genoms sowie der Funktion und Regulation genomischer Elemente in der veterinärmedizinischen Forschung. Dabei setzen wir moderne OMICs-Technologien, inklusive RNA-seq, ATAC-seq und Hi-C, sowie DNA-Sprachmodelle ein, um regulatorische Muster zu entschlüsseln und komplexe Genotyp-Phänotyp-Zusammenhänge besser zu verstehen. Im Fokus stehen dabei Prozesse des Wachstums und der Skelettentwicklung, insbesondere an Wachstumsfugen, in Haus- und Nutztieren wie zB. dem Schwein. Wir untersuchen zudem die Rolle von Stammzellen in Regeneration und Degeneration von Geweben sowie funktionelle und epigenetische Ursachen von Erkrankungen, die durch Umweltfaktoren wie Infektionen oder Temperatureinflüsse beeinflusst werden. Ein weiterer Schwerpunkt liegt auf der Analyse evolutionärer Prozesse bei Haus- und Nutztieren, insbesondere im Hinblick auf Selektionssignaturen und die genomischen Folgen künstlicher Selektion. Unsere Arbeiten integrieren in vitro-Modelle mit Einzelzellanalysen und bioinformatischen Methoden, um funktionelle Mechanismen auf molekularer Ebene aufzuklären.
AG-Leiterin:
Forschungsprojekte
Laufende Projekte
Aktuelle Forschungsprojekte (Auswahl; DFG-Projekte: siehe DFG GEPRIS) in der funktionellen Genomik

Wachstum & Krankheit
- MEASURE: Multi-omics Studien zur Körpergröße im Tiermodell – die genetische Architektur der Körpergröße des Schweines (DFG-Heisenberg-Studie)
- 3D-DRIVE: Dynamische 3D-Genom Reorganisation des porcinen Wachstums
- SKIP: Funktionelle Analyse von skelettmodifizierenden Genen und interkativen Genexpressions-Netzwerken

- SPAID-Forschung: Funktionelle Studien zur Shar Pei Autoinflammatorischen Erkrankung (siehe hier)

Genomstruktur & Evolution
- WORMICs: 3D Genomics des Mehlwurms
-
Strukturelle Variation im Zusammenhang mit Domestikation und Selektion von Haustierrassen
Eine vollständige Liste der drittmittelbasierten Forschungsprojekte finden sie in der TiHo-Datenbank für Forschungsprojekte
Publikationen
Ausgewählte Publikationen
Genom & Selektionssignaturen:
- Berghöfer J, Khaveh N, Mundlos S, Metzger J (2022) Simultaneous testing of rule- and model-based approaches for runs of homozygosity detection opens up a window into genomic footprints of selection in pigs. BMC Genomics 23 (1): 1-26. https://doi.org/10.1186/s12864-022-08801-4 (open access)
Wachstumsdefizienzen & Miniaturisierung:
- Halecker, S., Metzger, J., Strube, C., Krabben, L., Kaufer, B. & Denner, J. (2021) Virological and Parasitological Characterization of Mini-LEWE Minipigs Using Improved Screening Methods and an Overview of Data on Various Minipig Breeds. Microorganisms 9, 9122617. https://doi.org/10.3390/microorganisms9122617
- Schachler K, Minx J-O, Sürie C, Distl O, Metzger J. (2020) Genetic characterisation of Mini-LEWE as resource population for experimental research. Berliner und Münchener Tierärztliche Wochenschrift 133, DOI: 10.2376/1439-0299-2020-15
- Metzger J, Rau J, Naccache F, Bas Conn L, Lindgren G, Distl O. (2018) Genome data uncover four synergistic key regulators for extremely small body size in horses. BMC Genomics 19(1):492. https://doi.org/10.1186/s12864-018-4877-5 (open access)
- Metzger J, Gast AC, Schrimpf R, Rau J, Eikelberg D, Beineke A, Hellige M, Distl O (2017) Whole-genome sequencing reveals a potential causal mutation for dwarfism in the Miniature Shetland pony. Mammalian Genome 28, 143-151. https://doi.org/10.1007/s00335-016-9673-4
- Metzger J, Distl O (2020) Book section: Genetics of Equine Orthopedic Disease. Veterinary Clinics of North America: Equine Practice, 36 (2): 289-301, Edited by Carrie J. Finno. https://doi.org/10.1016/j.cveq.2020.03.008
Transcriptom und Multi-OMICs Studien in Schwein & anderen Haustieren:
- Khaveh N, Buschow R, Metzger J (2024) Deciphering transcriptome patterns in porcine mesenchymal stem cells promoting phenotypic maintenance and differentiation by key driver genes. Frontiers in Cell and Developmental Biology, 12: 1478757. https://doi.org/10.3389/fcell.2024.1478757
- Khaveh N, Schachler K, Berghöfer J, Jung K, Metzger J (2023) Altered hair root gene expression profiles highlight calcium signaling and lipid metabolism pathways to be associated with curly hair initiation and maintenance in Mangalitza pigs. Frontiers in Genetics, 14: 1184015. https://doi.org/10.3389/fgene.2023.1184015 (open access)
- Schachler K, Distl O, Metzger J (2020) Tracing selection signatures in the pig genome gives evidence for selective pressures on a unique curly hair phenotype in Mangalitza. Scientific Reports, 10 (1): 1-12. https://doi.org/10.1038/s41598-020-79037-z (open access)
- Metzger, J, Schrimpf, R, Philipp, U & Distl, O (2013) Expression Levels of LCORL Are Associated with Body Size in Horses. PLoS ONE 8, https://doi.org/10.1371/journal.pone.0056497 (open access)
Für weitere Publikationen, siehe Google Scholar
Karrieremöglichkeiten
Wir sind immer auf der Suche nach hochmotivierten Wissenschaftlern mit Interesse/Expertise im Bereich Genetik, Genomik, Stammzellentwicklung und bioinformatische Methoden.
Bei Interesse schicken sie bitte eine Bewerbung (Anschreiben, Lebenslauf, Empfehlungsschreiben von zwei Mentoren) als ein PDF an: julia.metzger@tiho-hannover.de




