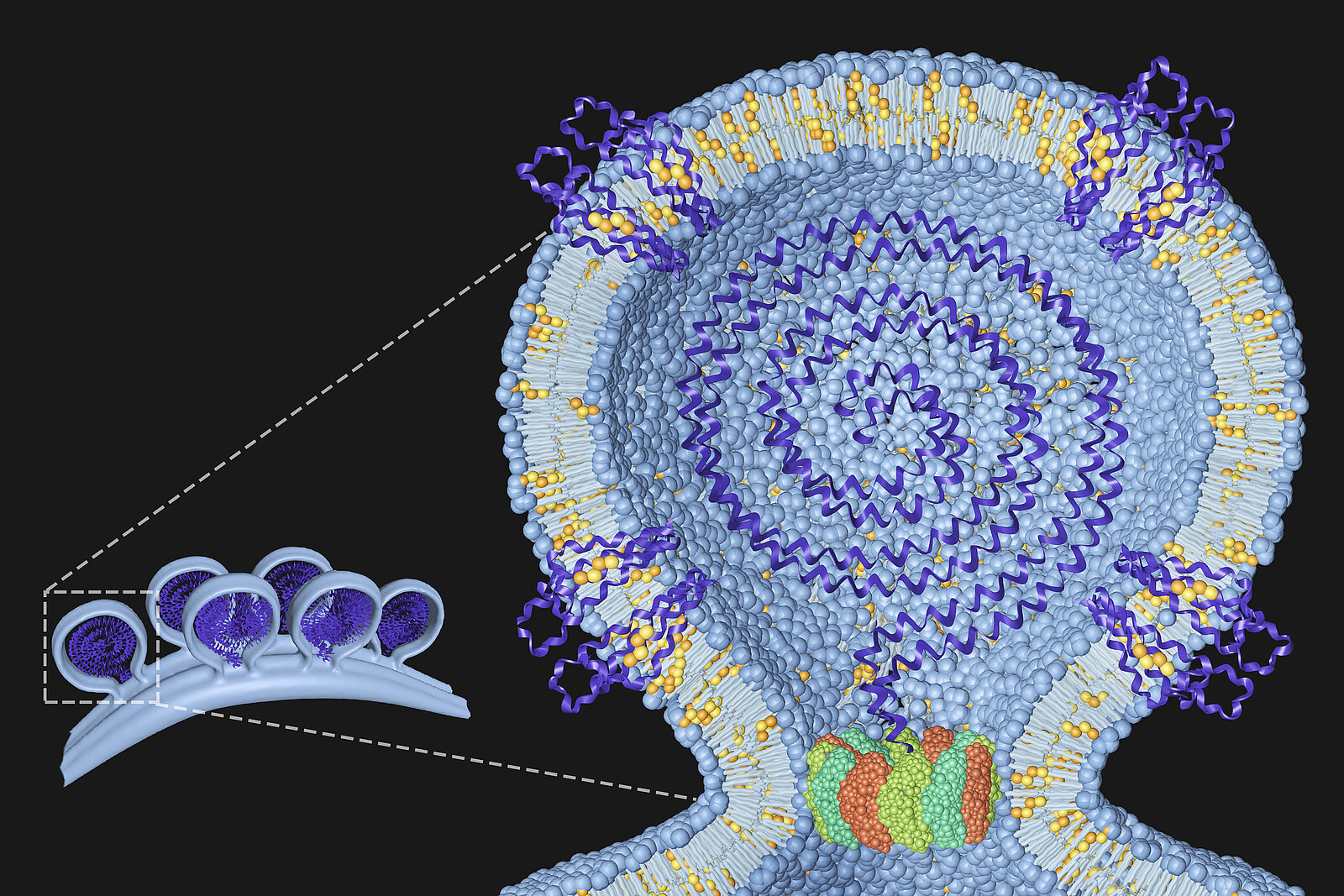
Unsere Gruppe ist daran interessiert, die Protein- und Lipiddeterminanten von Virusinfektionen zu verstehen. Die Abbildung zeigt ein Modell des Replikationskomplexes des Chikungunyavirus an der Plasmamembran mit der viralen RNS in der Mitte und von uns identifizierten Wirtsproteinen und Lipiden, die in die Membran eingebettet sind. Der dodekamere Ring am Hals stellt das nicht strukturelle Protein 1 (nsP1) des Chikungunyavirus dar. Details sind Lasswitz et al., mBio 2022 zu entnehmen.
Aktuelle Projekte der AG Gerold
14
Ergebnisse.
Verständnis grundlegender Mechanismen der Membranverformbarkeit von Insektenzellen
Understanding fundamental mechanisms governing insect cell membrane
deformability
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Oktober 2023 bis September 2026
Entwicklung eines ex-vivo-Modells zur Untersuchung der zoonotischen Übertragung von Noroviren
Development of an ex vivo model to study the zoonotic transmission of
noroviruses
Projektverantwortliche: Prof. Dr. Gisa Gerold; Dr. Nele Villabruna
Laufzeit: Mai 2023 bis Oktober 2024
Niedersachsen KONSORT: Evolutionärer Rüstungswettlauf zwischen Arboviren und ihren Wirten
Niedersachsen KONSORT: Evolutionary Arms Race of Arboviruses and their Hosts
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Februar 2023 bis April 2024
Modell zur Arbovirus-Infektion der Haut - Mozart
Model for arbovirus infection of the skin - Mozart
Projektverantwortliche: Prof. Dr. Gisa Gerold; Dr. Cora Stegmann; Prof. Dr. Stefanie Becker; Dr. Fanny Hellhammer
Laufzeit: Oktober 2023 bis Dezember 2024
Identifizierung und Charakterisierung von Wirtsfaktoren für Alphaviren, die den Tropismus von menschlichem Gewebe bestimmen
Identification and characterization of alphavirus host factors determining human tissue tropism
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Januar 2022 bis Dezember 2024
Verständnis der Rolle von Phosphatidylserin und seinen Rezeptoren bei der artenübergreifenden Übertragung von Alphaviren
Understanding the role of phosphatidylserine and its receptors in cross-species transmission of alphaviruses
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Oktober 2021 bis September 2024
Big Data in den Lebenswissenschaften der Zukunft: Paving the Way towards Personalized Prevention and Care of Severe Norovirus Gastroenteritis (PRESENt)
Big Data in Life Sciences: Paving the Way towards Personalized Prevention and Care of Severe Norovirus Gastroenteritis (PRESENt)
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Januar 2020 bis September 2023
Fast Track COFONI: Air-Liquid-Interface-Kulturen von menschlichem primärem distalem Atemwegsepithel für die In-vitro-Modellierung von SARS-CoV2-Infektionen
Fast Track COFONI: Air Liquid Interface cultures of human primary distal respiratory epithelial for in vitro modelling of SARS-CoV2 infections
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: August 2021 bis Juli 2022
Bewerbung für Challenge - Antivirale Wirkstoffe CRISPR/Cas13-vermittelte antivirale Therapie
Application for Challenge ? Antiviral Agents CRISPR/Cas13-mediated antiviral therapy
Projektverantwortliche: Prof. Dr. Albert Osterhaus; Prof. Dr. Gisa Gerold
Laufzeit: November 2021 bis Oktober 2022
COVID-Protect - Wirksamkeit von Nrf2 Aktivatoren für die Verhinderung von Zellschädigung bei COVID-19 - Teilprojekt Toxizitäts- und Wirksamkeitsstudien
COVID-Protect - Efficacy of Nrf2 activators for preventing cell injury in COVID-19 - Subproject toxicity and efficacy studies
Projektverantwortliche: Prof. Dr. Gisa Gerold
Laufzeit: Juli 2020 bis Juni 2022




