Das Humane Respiratorische Syncytial-Virus (RSV) ist weltweit der häufigste Auslöser akuter Atemwegsinfektionen bei Säuglingen und Kleinkindern. Forschende des Research Center for Emerging Infections and Zoonoses (RIZ) der Stiftung Tierärztliche Hochschule Hannover (TiHo) stellten in der hochrangigen Fachzeitschrift PNAS gemeinsam mit Wissenschaftlerinnen und Wissenschaftlern der Medizinischen Hochschule Hannover (MHH) eine neue Untersuchungsmethode für aktuell zirkulierende Stämme des Respiratorischen Synzytial-Virus vor.
Die Ausgangslage
Ältere Kinder und Erwachsene entwickeln bei einer RSV-Infektion in aller Regel nur leichte, erkältungsähnliche Symptome. Bei kleinen Kindern bis zu einem Alter von fünf Jahren, bei Personen mit einem geschwächten Immunsystem und bei älteren Menschen hingegen kann eine RSV-Infektion zu einer schweren Lungenentzündung und in einigen Fällen zum Tod führen. Die Infektion tritt vor allem in den Wintermonaten und im Frühjahr auf.
Obwohl RSV bereits 1956 entdeckt wurde, gibt es bis heute keine zugelassenen Impfstoffe oder wirksame Medikamente. Die einzige verfügbare Therapie (Palivizumab) ist eine passive Impfung, die auf monoklonalen Antikörpern basiert, welche gegen Proteine auf der Hülle des RS-Virus gerichtet sind. Die Therapie kann bei besonders gefährdeten Kleinkindern eingesetzt werden, um schweren Krankheitsverläufen vorzubeugen. Sie erhalten die Antikörper während der von November bis April dauernden RSV-Saison monatlich per Spritze in den Muskel.
Obwohl weltweit die beiden Hauptuntertypen A und B zirkulieren, konzentrierte sich die Forschung in den vergangenen 60 Jahren hauptsächlich auf RSV-A-Stämme. Diese Stämme werden seit Jahrzehnten im Labor eingesetzt und haben sich entsprechen adaptiert: Sie weisen im Vergleich zu den in der Bevölkerung zirkulierenden Virusstämmen große Unterschiede im Viruswachstum, der Zellfusion und hinsichtlich der thermischen Stabilität auf. Mit diesen Stämmen lassen sich folglich keine umfangreichen Aussagen über die Beschaffenheit aktueller RS-Virusstämme erlangen.
Die neue Methode
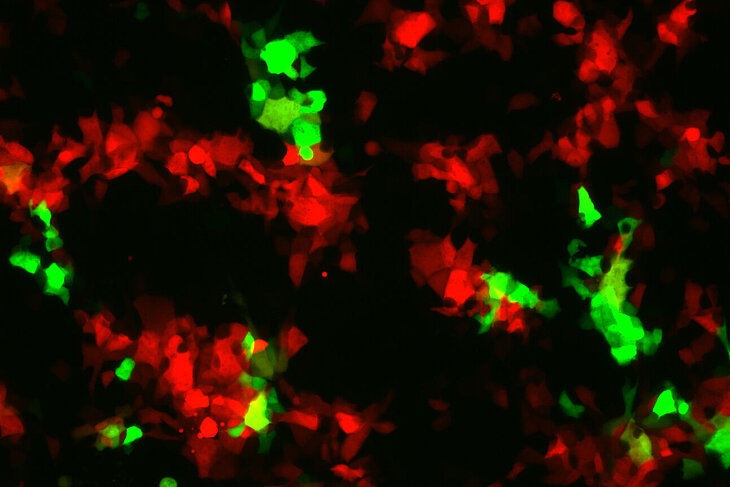
Ein Forschungsteam unter der Leitung von Dr. Martin Ludlow, Projektleiter für Virologie im RIZ, und RIZ-Direktor, Professor Dr. Albert Osterhaus, entwickelte deshalb eine Methode, die es ermöglicht, die Einflüsse von Veränderungen in der Erbinformation aktueller RSV-A- und B-Subtypen zu untersuchen. Die hierfür benutzten Virusstämme stammten jeweils direkt von erkrankten Patienten. Das Forschungsteam übertrug die gesamte Erbinformation der jeweiligen Subtypen, die sich beim RSV auf einem RNA-Einzelstrang befindet, in eine sogenannte complementary DNA, cDNA. Mit Hilfe dieser cDNA konnte die genetische Information des Virus in einen sogenannten Klonierungsvektor (Plasmide) übertragen werden. Diese Umwandlung ermöglichte es ihnen, das virale Erbgut stabil zu reproduzieren und außerdem spezifisch zu verändern. Auf diese Weise konnten sie Mutationen, die die Viren weniger anfällig für den monoklonalen Antikörper Palivizumab werden lassen, direkt in das virale Genom integrieren und deren Einfluss auf die virale Fitness und die Virus-Wirt-Beziehungen gezielt untersuchen. Des Weiteren machte es diese Technologie möglich, als Markierung fluoreszierende oder lumineszierende Proteine in das virale Erbgut zu integrieren.
Aus den so erstellten Plasmiden, welche die manipulierte Erbinformation des Virus enthalten, erzeugte das Forschungsteam wiederum so genannte rekombinante Viren. Fluoreszierend oder lumineszierend markierte Viren sind im Labor ein wichtiges Hilfsmittel, um die Viren im Versuch schneller und effektiver nachweisen können. Diese rekombinanten Viren können nun genutzt werden, um neue Therapieansätze zu testen oder um neue Erkenntnisse über den Weg des Virus in die Zelle zu gewinnen.
„Mit unserem neuen Ansatz haben wir eine stabile, zuverlässige und einfache Methode, um RSV-Varianten zu untersuchen. Und mit den fluoreszierenden Viren können wir beispielsweise die für die Infektion relevanten Virus- und Wirtsfaktoren untersuchen“, erklärt Ludlow. Osterhaus ergänzt: „Dieses neue Reverse-Genetik-System ermöglicht Studien in enger Zusammenarbeit mit Pharmaunternehmen, um neue therapeutische Antikörper, Virostatika und Impfstoffe zu entwickeln. Wir werden solche Untersuchungen mit In-vitro- und Ex-vivo-Versuchsmodellen kombinieren. Sie können teilweise unser derzeitiges RSV-Frettchenmodell ersetzen“, sagt Osterhaus.
Die Studie wurde finanziert durch Fördergelder der EU, Deutschlands und des Landes Niedersachsen.
Die Originalveröffentlichung
Reverse genetics systems for contemporary isolates of respiratory syncytial virus enable rapid evaluation of antibody escape mutants
Jo WK, Schadenhofer A, Habierski A, Kaiser FK, Saletti G, Ganzenmueller T, Hage E, Haid S, Pietschmann T, Hansen G, Schulz TF, Rimmelzwaan GF, Osterhaus ADME, Ludlow M (2021)
PNAS, DOI doi.org/10.1073/pnas.2026558118
Kontakt
Dr. Martin Ludlow
Stiftung Tierärztliche Hochschule Hannover
Research Center for Emerging Infections and Zoonoses (RIZ)
Tel.: +49 511 953-6112
martin.ludlow@tiho-hannover.de
Prof. Dr. Albert Osterhaus
Stiftung Tierärztliche Hochschule Hannover
Research Center for Emerging Infections and Zoonoses (RIZ)
Tel.: +49 511 953-6140
albert.osterhaus@tiho-hannover.de